NGS测序软件模块 SEQNEXT实验室分析捕获
添加到我的收藏夹
添加到产品对比表
产品规格型号
- 应用
- 实验室, NGS测序
- 功能
- 分析, 捕获, 染色体图谱绘制
产品介绍
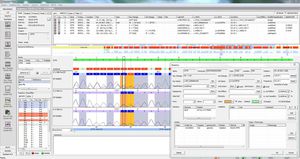
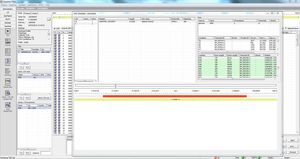
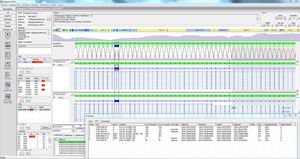
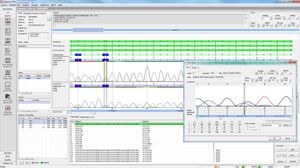
SEQNEXT是一个强大的、用户友好的应用程序,用于下一代测序数据的绘图、比对和变体检测。SEQNEXT可以分析来自所有常见测序平台和试剂盒(基于PCR的、富集的、序列捕获的)的数据。所有检测到的变体,如任何长度的缺失、插入和缩进、SNPs、重复区域以及CNVs和基因融合的可视化是清晰和直观的。
SEQNEXT提供对公共SNP数据库的访问,如dbSNP、1000 Genomes、ClinVar、ExAC和gnomAD的分类和过滤。所有的结果数据都可以与我们的变异数据库进行交流,以分享经验和知识--varSEAK,转移到实验室内部的LIM系统和/或作为个性化的病人报告发布。
新改进的病毒测序数据分析,如SARS-CoV-2
三重分析和改进的集合分析
变异体表中的序列本体注释
跳转到varSEAK和所有可用的外部数据库,如dbSNP、ClinVar、UK10K、ESP、gnomAD和ExAC。
全外显子组测序(WES)分析
与所有常见的下一代测序平台和试剂盒的数据兼容
通过导入基于hg19和/或hg38的床单或清单文件,轻松设置单个目标区域(ROI)。
导入基因列表以过滤和分析WES数据
分析fastq-、fastq.gz-或bam-文件
高效的绘图、比对、质量和变体调用标准(使用的算法:BWA和Smith-Waterman,由JSI改编)。
分析和变体调用的个性化设置(≥ 0.1 %)。
对任何长度的缺失、插入和缩减、SNPs、重复区域以及CNVs和基因融合的检测具有高灵敏度和特异性
---